Contents
Was ist Sequenzausrichtung?
Ein Sequenz-Alignment ist eine Möglichkeit, die Sequenzen von DNA, RNA oder Protein anzuordnen, um Ähnlichkeitsbereiche zu identifizieren, die eine Folge funktioneller, struktureller oder evolutionärer Beziehungen zwischen den Sequenzen sein können. Ausgerichtete Sequenzen von Nukleotid- oder Aminosäureresten werden typischerweise als Reihen innerhalb einer Matrix dargestellt. Zwischen den Resten werden Lücken eingefügt, damit gleiche oder ähnliche Zeichen in aufeinanderfolgenden Spalten ausgerichtet werden. Sequenzausrichtungen werden auch für nicht-biologische Sequenzen verwendet, wie zum Beispiel die Berechnung der Distanzkosten zwischen Strings in einer natürlichen Sprache oder in Finanzdaten.
Globale Sequenzausrichtung
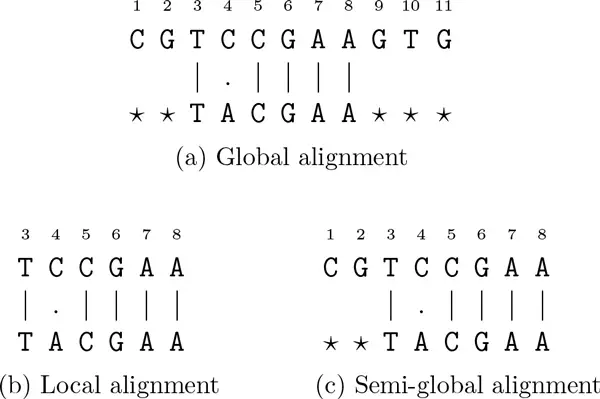
Globales Alignment wird verwendet, um Sequenzen in Fällen zu vergleichen, in denen wir Grund zu der Annahme haben, dass die Sequenzen entlang ihrer gesamten Länge verwandt sind. Wenn beispielsweise die Sequenzen s und t zwei unabhängige Sequenzierungsläufe desselben PCR-Produkts sind, sollten sie sich nur in Positionen unterscheiden, an denen Sequenzierungsfehler vorliegen. Um diese Sequenzierungsfehler zu finden, richten wir die gesamte Sequenz s mit der gesamten Sequenz t aus . Andere Anwendungen des globalen Alignments umfassen das Auffinden von Mutationen in eng verwandten Gen- oder Proteinsequenzen und die Identifizierung von Einzelnukleotidpolymorphismen (SNPs).
Beispiele für globale Ausrichtungswerkzeuge sind:
- EMBOSS-Nadel
- Needleman-Wunsch Global Align Nukleotidsequenzen (spezialisierte BLAST)
Was Sie über die lokale Sequenzausrichtung wissen müssen
- Beim globalen Alignment wird versucht, die gesamte Sequenz auszurichten (End-to-End-Alignment).
- Ein globales Alignment enthält alle Buchstaben sowohl aus der Abfrage- als auch aus der Zielsequenz.
- Wenn zwei Sequenzen ungefähr gleich lang und ziemlich ähnlich sind, eignen sie sich für ein globales Alignment.
- Geeignet zum Alignment zweier eng verwandter Sequenzen.
- Globale Alignments werden normalerweise durchgeführt, um homologe Gene zu vergleichen, z.
- Eine allgemeine globale Ausrichtungstechnik ist der Needleman-Wunsch-Algorithmus.
Lokale Sequenzausrichtung
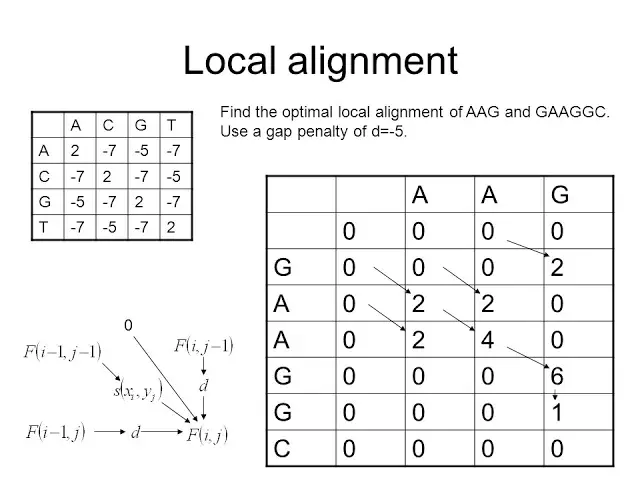
Local Alignment befasst sich mit Fällen, in denen wir nur erwarten, isolierte Regionen mit Ähnlichkeit zu finden. Ein Beispiel ist das Alignment von genomischer DNA stromaufwärts von zwei co-exprimierten Genen, um konservierte Regionen zu finden, die Transkriptionsfaktor-Bindungsstellen entsprechen können. Eine andere Anwendung ist die Identifizierung von konservierten Domänen 1 in zwei Aminosäuresequenzen, die Proteine codieren, die eine oder mehrere Domänen teilen, aber ansonsten nicht verwandt sind.
Beispiele für lokale Ausrichtungswerkzeuge sind:
- SPRENGEN
- EMBOSS Wasser
- LALIGN
Was Sie über die lokale Sequenzausrichtung wissen müssen
- Findet lokale Regionen mit der höchsten Ähnlichkeit zwischen den beiden Sequenzen.
- Ein lokales Alignment richtet eine Teilzeichenfolge der Abfragesequenz an einer Teilzeichenfolge der Zielsequenz aus.
- Beliebige zwei Sequenzen können lokal ausgerichtet werden, da lokale Ausrichtung Abschnitte von Sequenzen mit hohem Übereinstimmungsgrad findet, ohne die Ausrichtung der restlichen Sequenzregionen zu berücksichtigen.
- Geeignet zum Ausrichten von divergenteren Sequenzen oder entfernt verwandten Sequenzen.
- Wird verwendet, um konservierte Muster in DNA-Sequenzen oder konservierte Domänen oder Motive in zwei Proteinen herauszufinden.
- Ein allgemeines lokales Ausrichtungsverfahren ist der Smith-Waterman-Algorithmus.
Lesen Sie auch: Unterschied zwischen Replikation und Transkription
Unterschied zwischen globaler und lokaler Sequenzausrichtung in Tabellenform
| VERGLEICHSGRUNDLAGE | GLOBALE SEQUENZAUSRICHTUNG | LOKALE SEQUENZAUSRICHTUNG |
| Beschreibung | Beim globalen Alignment wird versucht, die gesamte Sequenz auszurichten (End-to-End-Alignment). | Findet lokale Regionen mit der höchsten Ähnlichkeit zwischen den beiden Sequenzen. |
| Beispiele für Werkzeuge | -EMBOSS-Nadel -Needleman-Wunsch Global Align Nukleotidsequenzen (spezialisierte BLAST) | -BLAST -EMBOSS Water -LALIGN |
| Funktion | Ein globales Alignment enthält alle Buchstaben sowohl aus der Abfrage- als auch aus der Zielsequenz. | Ein lokales Alignment richtet eine Teilzeichenfolge der Abfragesequenz an einer Teilzeichenfolge der Zielsequenz aus. |
| Zwei Sequenzen | Wenn zwei Sequenzen ungefähr gleich lang und ziemlich ähnlich sind, eignen sie sich für ein globales Alignment. | Beliebige zwei Sequenzen können lokal ausgerichtet werden, da lokale Ausrichtung Abschnitte von Sequenzen mit hohem Übereinstimmungsgrad findet, ohne die Ausrichtung der restlichen Sequenzregionen zu berücksichtigen. |
| Eignung | Geeignet zum Alignment zweier eng verwandter Sequenzen. | Geeignet zum Ausrichten von divergenteren Sequenzen oder entfernt verwandten Sequenzen. |
| Benutzen | Globale Alignments werden normalerweise durchgeführt, um homologe Gene zu vergleichen, z. | Wird verwendet, um konservierte Muster in DNA-Sequenzen oder konservierte Domänen oder Motive in zwei Proteinen herauszufinden. |
| Allgemeine Technik | Eine allgemeine globale Ausrichtungstechnik ist der Needleman-Wunsch-Algorithmus. | Ein allgemeines lokales Ausrichtungsverfahren ist der Smith-Waterman-Algorithmus. |
